三维结构信息预测蛋白质相互作用,极为准确蛋白质间的作用机理。近日,同济大学电子与信息工程学院计算机科学与技术系关佶红教授指导的博士生邓磊与美国哥伦比亚大学的张强峰博士、Barry Honig教授等人合作撰写的论文“基于结构的全基因组蛋白质相互作用预测”在国际顶级学术期刊《自然》杂志上在线发表(doi:10.1038/nature11503)。

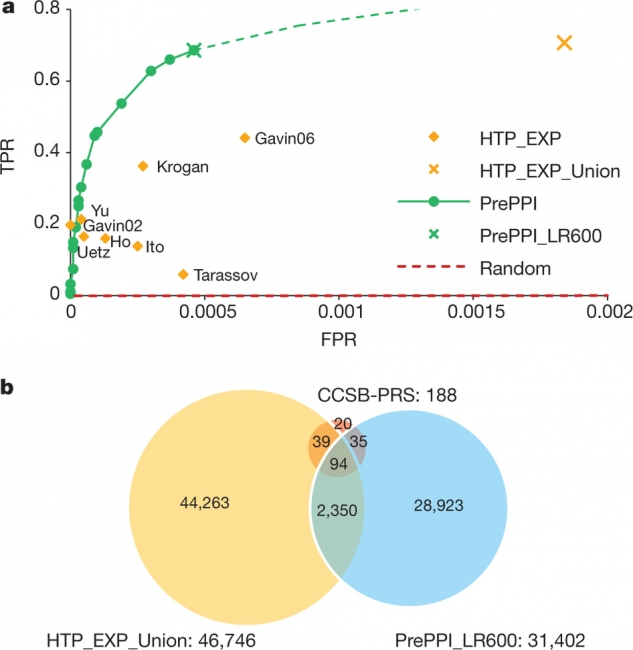
蛋白质相互作用研究能够从分子水平上揭示蛋白质的功能,帮助揭示生长发育、新陈代谢、分化和凋亡等细胞活动的规律。在全基因组范围内识别蛋白质相互作用对是解释细胞调控机制的重要一步。随着蛋白质相互作用实验技术的发展,人们能够获得大量的蛋白质相互作用数据,甚至能够在全基因组范围内对蛋白质相互作用进行分析。然而,由于实验技术的限制,很多高通量实验方法测得的蛋白质相互作用数据的错误率都比较高。并且,由于待测的蛋白质对数目巨大,若要对其逐一验证,需要花费大量的人力和物力。该论文提出了一种基于三维结构信息的全基因组蛋白质相互作用计算预测方法----PrePPI。研究结果表明基于结构信息的相互作用预测方法无论是在准确率还是在覆盖度上均大大优于基于非结构信息的方法。使用三维结构信息在预测相互作用方面所取得的惊人的效果,归功于大量使用了同源模型以及结合了蛋白质之间较近和较远的几何关系。此外,PrePPI还集成了其它多种功能线索,其预测结果甚至可以与高通量实验相媲美。
邓磊为我校电信学院计算机系2008级博士生,于2010-2011年在美国哥伦比亚大学公派联合培养期间在Barry Honig教授实验室作为主要研究人员参与了该项研究。Barry Honig是美国哥伦比亚大学计算生物与生物信息中心主任、教授,美国科学院院士,霍华德•休斯医学研究所(HHMI)研究员,PNAS等著名国际期刊编辑。